| Autor | Institution |
|---|---|
| Jennifer Schneiderbanger | TUM School of Life Science |
| Datum | 18. März 2021 |
| Ausgabe | 1 |
| Jahrgang | 89 |
| Seitenzahl | 26-30 |
Die Hürden des Bieres
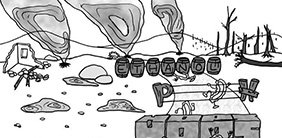
Die sogenannte ‚Hürdentheorie‘ wurde durch Leistner postuliert [1] und wird seither oft zitiert, um zu verdeutlichen, dass Mikroorganismen mehrere Hindernisse überwinden müssen, um im Medium Bier zu wachsen. Diese in ihrer antimikrobiellen Stärke variierenden Herausforderungen sind in Abbildung 1 illustriert.

Als intrinsische Hürden werden Bierinhaltsstoffe oder Biereigenschaften bezeichnet, die einen inhibierenden Effekt auf vorhandene Mikroorganismen haben. Tabelle 1 stellt diese intrinsischen Hürden zusammen mit ihren durchschnittlich in Bier vorkommenden Konzentrationen und deren Auswirkung(en) auf Mikroorganismen dar. Dabei ist zu berücksichtigen, dass sich die antimikrobiellen Aktivitäten des Hopfens nur gegen Gram-positive Mikroorganismen richten. In einer Studie von Fernandez & Simpson konnte belegt werden, dass die Sensibilität von Bier gegenüber einem mikrobiellen Verderb hauptursächlich mit dessen pH-Wert und der Konzentration an undissoziierten Hopfensäuren korreliert [3].
Als extrinsische Hürden werden diejenigen Prozessschritte während der Bierproduktion bezeichnet, die zusätzlich zu den intrinsischen Faktoren eine weitere Schädigung des Produktes durch Mikroorganismen eindämmen (Beispiele: Würzekochung, Pasteurisierung, Filtration).
Die über die Zugabe von Hopfen eingebrachten alpha-Säuren und deren hitzeisomerisierte iso-alpha-Formen gelten als die wichtigsten Bestandteile von Bier im Hinblick auf die mikrobiologische Stabilität [4, 13]. Ihre antimikrobielle Wirkung auf Gram-positive Mikroorganismen zeigt sich auf verschiedene Weise: (1) Isomerisierte Hopfensäuren wirken als Protonen-Ionophore, die nach der Überwindung der Zellmembran in ihrer undissoziierten Form im Zellinnern deprotonieren und so das Zellinnere ansäuern, wodurch der pmf-abhängige Nährstoffimport, die Atmung, DNA- und RNA-Synthese sowie die Aktivität essenzieller Enzyme inhibiert werden [4, 11, 12]; (2) sie beeinflussen transmembrane Redoxreaktionen, was in oxidativem Stress für die Zelle resultiert [14]; (3) in die Zelle eingedrungene Hopfensäuren komplexieren mit Mn2+-Ionen, deren Verlust zu Problemen in Energiegewinnung und Aufrechterhaltung der Redox-Homöostase und auch zum Verlust der Cofaktoren essenzieller Enzyme führt [12, 13, 15].
Bierschädliche Bakterien
Für den Verderb von Bier werden hauptsächlich Bakterien verantwortlich gemacht, insbesondere Vertreter der Gram-positiven Milchsäurebakterien-Gattungen Lactobacillus (L.) und Pediococcus (P.) [16]. Innerhalb dieser Gattungen wiederum sind zahlreiche Spezies dazu in der Lage, Bier zu verderben. Mit Abstand am häufigsten in kontaminierten Proben anzutreffen ist die Spezies Lactobacillus brevis, die aufgrund ihrer Dominanz auch im Fokus vieler Studien über ihre genetische Ausstattung steht [17-24]. Aus der Gattung Pediococcus ist die am häufigsten vorkommende und demnach auch auf genetischer Ebene analysierte Art P. damnosus [25-27].

Ein Überblick über die als Primär- oder Sekundärkontaminanten in der Brauerei vorkommenden Milchsäurebakterien ist in Tabelle 2 gegeben.
Des Weiteren muss angemerkt werden, dass die Fähigkeit zum Bierverderb tendenziell stammabhängig und in den meisten Fällen nicht speziesabhängig ist. Bei Spezies, die bisher nur aus der Bierumgebung isoliert wurden (z.B. L. backii, L. lindneri), reicht meist eine Identifizierung auf Artebene, um einen Rückschluss auf die Bierschädlichkeit des Isolats ziehen zu können. Innerhalb anderer Spezies dagegen (Beispiel: L. brevis) gibt es bierschädliche und nicht-bierschädliche Stämme, weswegen aus der alleinigen Artidentifizierung kein Rückschluss gezogen werden kann.
Auf Basis dieser Erkenntnis wird seit vielen Jahren versucht, eine spezies-unabhängige Methode zu entwickeln, mit der schnell und zuverlässig auf die Bierschädlichkeit eines Isolats geschlossen werden kann. Im Zuge der zunehmend genombasierten Analytik versuchten gerade im letzten Jahrzehnt zahlreiche Wissenschaftler, den Genen auf die Spur zu kommen, deren Anwesenheit (alleine oder in Kombination mit anderen Genen) ausschlaggebend für die Fähigkeit zum Bierverderb ist. In diversen Studien wurde bereits ermittelt, dass das Potential zur Bierschädigung multifaktoriell ist und meist nicht auf die Anwesenheit eines einzigen Gens zurückzuführen ist [13, 17, 25, 30-35].
Die Gene als Grundlage der Resistenzmechanismen
Die Gene, die mit dem Potential zum Bierverderb in Verbindung gebracht werden, sind meist auf mobilen genetischen Elementen lokalisiert, die durch horizontalen Gentransfer unter anderem zwischen verschiedenen Spezies ausgetauscht werden können [36, 37]. Einige der im Folgenden aufgeführten Gene wurden nur in einer einzigen Spezies als valider Indikator für die Ausprägung eines bierschädigenden Potentials beschrieben, andere in zahlreichen Spezies.
horA
Anfänglich wurde von Sami et al. ein Gen beschrieben, das bierschädlichen Bakterien einen Überlebensvorteil im unwirtlichen Biermilieu schaffen sollte [38, 39]. horA ist plasmid-lokalisiert und kodiert für einen sogenannten Multidrug-Transporter der ATP-binding-casette-Familie, der in der Lage ist, unter Aufwendung von Energie in Form von ATP schädliche Hopfensäuren aus dem Zellinneren auszuschleusen [23]. Haakensen et al. beschrieben horA als das einzige Gen, dessen Anwesenheit in Milchsäurebakterien Rückschlüsse auf bierschädliches Potential zulässt [40]. Ein solches Gen, das speziesübergreifend zwischen bierschädlichen und nicht-bierschädlichen Isolaten unterscheiden kann, wird genetischer Marker genannt [41, 42]. Da allerdings auch Isolate, die das horA-tragende Plasmid verloren hatten, ein gewisses Bierschädigungspotential aufweisen können, wurden zusätzliche Resistenzmechanismen vermutet [30, 41].
horC / horB
Iijima et al. beschrieben die pmf-abhängige Multidrug-Efflux-Pumpe horC, die ein weiterer genetischer Marker für die Fähigkeit zum Bierverderb ist [21]. horC ist plasmid-lokalisiert und wird durch horB reguliert, wobei die hochkonservierte Sequenz von horB darauf hindeutet, dass es essenziell für die Transkription von horC ist [21, 34, 43, 44]. horC wird wie horA als aktiver Hopfenresistenzmechanismus beschrieben, der einen reduzierten Netto-Eintrag von undissoziierten Hopfensäuren in das Cytoplasma bewirkt. Beide mit der Hopfentoleranz assoziierten Gene lassen sich (nahezu) exklusiv in bierschädlichen Bakterien finden [37]. Da die beiden Gene immer gleichzeitig mit anderen, flankierenden open reading frames (ORF) abgelesen werden, die zum Teil in die Zellwandsynthese involviert sind, ist es möglich, dass nicht nur horA und horC, sondern das gesamte Gencluster (siehe Abbildung 2) die gesteigerte Hopfentoleranz vermittet [41].
Suzuki et al. beschrieben, dass durch die Kombination von horA und horC alle Lactobacillus-Isolate mit bierschädigendem Potential detektiert werden konnten [43]. Im Falle von Pediococcus-Isolaten ist die Aussagekraft dieser beiden Gene deutlich geringer [32, 36, 40]. In späteren Studien allerdings konnte festgestellt werden, dass horA und horC in manchen bierschädlichen Isolaten fehlten oder in manchen nicht-bierschädlichen Isolaten detektiert werden konnten [13, 32, 40]. Neben ihrer Rolle als hopfen-assoziierte Protonen-Pumpen sollen horA und horC weitere Funktionen in Bezug auf die Stressantwort in Bier erfüllen [37, 45]. Es ist außerdem anzumerken, dass der Verlust von horC im Vergleich zum Verlust von hitA oder horA die schwerwiegendste Auswirkung auf die Hopfentoleranz von L. brevis hat [19, 44].
hitA
Ein weiterer genetischer Marker wurde von Hayashi et al. beschrieben und als das Gen hitA identifiziert [31]. HitA kodiert für einen potentiellen pmf-abhängigen Multidrug-Transporter zweiwertiger Kationen, der durch die Anwesenheit von Hopfensäuren induziert wird und der schädlichen Aktivität der Hopfensäuren entgegenwirkt. HitA beeinflusst den intrazellulären Mn2+-Spiegel, indem der Gehalt an zweiwertigen Kationen in bierschädlichen Milchsäurebakterien aufrechterhalten bzw. durch Intrusion von Mn2+-Ionen in die Zelle stabilisiert wird [19, 37]. Bereits in frühen Studien wurde verdeutlicht, dass die Anwesenheit von hitA alleine nicht für die Ausprägung eines bierschädlichen Charakters verantwortlich sein kann [31].
Die Anwesenheit der hauptsächlich für die Ausprägung eines bierschädlichen Charakters verantwortlich gemachten genetischen Marker horA, horC und hitA alleine oder eine stabile Kombination daraus, lässt keine eindeutigen Rückschlüsse auf das Bierschädigungspotential eines Isolates zu [19]. Wahrscheinlicher ist, dass es weitere mit der Bierschädlichkeit in Zusammenhang stehende Gene gibt, deren Anwesenheit einem Isolat einen Überlebensvorteil in Bier sichert.
bsrA/bsrB
bsrA und bsrB sind chromosomal kodierte Multidrug-Transporter der ABC-Familie, die innerhalb der bierschädlichen Milchsäurebakterien nur in Pediococcen detektiert wurden und deren Anwesenheit mit dem Bierschädigungspotential korrelieren soll. Eine kombinierte Detektion der beiden Hopfentoleranz-assoziierten Genen bsrA und horA erzielte die beste Vorhersagbarkeit des Bierschädigungspotentials von Pediococcen [32].
mntB / mntC
Die Gene mntA, mntB und mntC zusammen formen ein komplettes Mn2+-Transport-System, deren Expressionsraten in L. casei in iso-alpha-Säure-haltigem Medium um ein Vielfaches anstiegen [46, 47]. Nach exogener Expression der Gene in einem nicht-bierschädlichen Lactococcus lactis-Stamm konnte verifiziert werden, dass mntB und mntC die Transportrate von Mn2+-Ionen ins Cytoplasma erhöhen und die Toleranz gegenüber Hopfensäuren signifikant steigern konnte [47].
fabZ
Das Gen fabZ aus einem hoch konservierten, plasmid-kodierten Fettsäurebiosynthese-Cluster wurde von Behr et al. als weiteres Markergen identifiziert [25]. Das Clwuster wurde innerhalb von bierschädlichen P. damnosus-, L. backii- und L. paucivorans-Isolaten detektiert, denen eine vollständige chromosomal-kodierte Fettsäurebiosynthese fehlte. Ohne fabZ konnten P. damnosus-Isolate in Bieren ohne Fettsäure-Zugabe nicht anwachsen [13].
mntH0274
Das chromosomale Gen mntH0274 soll für ein Mangan-Transportprotein in bierschädlichen Lactobacillus brevis-Stämmen kodieren und wird mit der Toleranz gegenüber niedrigen pH-Werten in Verbindung gebracht. Homologe Sequenzen lassen sich in allen veröffentlichten Komplettgenomen der Spezies L. brevis finden, aber nicht in anderen bierschädlichen Spezies wie beispielsweise in Pediococcen [48].
Weitere Gene
Das gtf-Gen, das für eine Glykosyltransferase kodiert, induziert den Aufbau einer Schutzschicht um LAB in alkoholischen Getränken herum und soll für eine erhöhte Alkohol-Toleranz sorgen [49]. In einer Studie, durchgeführt an 61 Isolaten, konnte jedoch festgestellt werden, dass die Anwesenheit des gtf-Gens weder mit erhöhter Bierschädlichkeit noch mit erhöhter Ethanoltoleranz korreliert [50]. Ein weiteres Gen, das für eine Glykosyltransferase kodiert (gtfD15), dagegen ermöglichte einem ansonsten unschädlichen Lactobacillus-Stamm das Wachstum in Bier [33].
Es wurde prognostiziert, dass das orf5-Gen für ein Transmembranprotein kodiert, das einem pmf-abhängigen Multidrug-Transporter ähnelt und mit der Extrusion von Hopfensäuren assoziiert sein könnte [51].
In einer Studie, die mit L. brevis-Isolaten durchgeführt wurde, wurden die chromosomalen Gene arsR (kodiert für einen Transkriptionsregulator) und cinA (kodiert für ein competence inducible protein) als Markergene für Bierschädigungspotential bestimmt [52]. Zwei weitere Transkriptionsregulatoren LysR und TetR konnten nur in bierschädlichen L. brevis-Isolaten detektiert werden. Ihre Rolle an der Antwort von Lactobacillen auf verschiedene Stressoren muss noch geklärt werden.
Weitere, bisher noch unbekannte Gene, die die Toleranz gegenüber den biereigenen Hürden erhöhen und so den bierschädlichen Milchsäurebakterien einen Überlebensvorteil sichern, scheinen wahrscheinlich [19]. Die Toleranz gegenüber der schädlichen Wirkung von Hopfensäuren steht dabei nach wie vor im Vordergrund. Allerdings wird immer wahrscheinlicher, dass die Kombination aus Hopfenresistenzmechanismen und weiteren plasmid-kodierten Proteinprodukten beispielsweise aus dem Bereich Membrantransport, Fettsäurebiosynthese oder Zellschutz durch die Bildung polymerer Substanzen synergistische Effekte in Bezug auf das Überleben in Bier hervorrufen könnten. y
LITERATUR
1. Leistner, L., Basic aspects of food preservation by hurdle technology. International Journal of Food Microbiology, 2000. 55(1-3): p. 181 - 186.
2. Menz, G. and F. Vriesekoop, Pathogens in beer, in Beer in health and disease prevention, V.R. Preedy and R.R. Watson, Editors. 2009, Elsevier Academic Press: Amsterdam.
3. Fernandez, J.L. and W.J. Simpson, Measurement and prediction of the susceptibility of lager beer to spoilage by lactic acid bacteria. Journal of Applied Microbiology, 1995. 78(4): p. 419 - 425.
4. Vriesekoop, F., et al., 125th anniversary review: Bacteria in brewing: The good, the bad and the ugly. Journal of the Institute of Brewing, 2012. 118(4): p. 335 - 345.
5. van de Guchte, M., et al., Stress responses in lactic acid bacteria. Antonie Van Leeuwenhoek International Journal of General and Molecular Microbiology, 2002. 82(1-4): p. 187 - 216.
6. Ingram, L.O., Ethanol tolerance in bacteria. Critical Reviews in Biotechnology, 1990. 9(4): p. 305 - 319.
7. Teuber, M., Schmalreck, A.F., MEMBRANE LEAKAGE IN BACILLUS-SUBTILIS 168 INDUCED BY HOP CONSTITUENTS LUPULONE, HUMULONE, ISOHUMULONE AND HUMULINIC ACID. Archiv Fur Mikrobiologie, 1973. 94(2): p. 159-171.
8. Behr, J., et al., Proteomic approach for characterization of hop-inducible proteins in Lactobacillus brevis. Applied and Environmental Microbiology, 2007. 73(10): p. 3300 - 3306.
9. Shimwell, J.L., On the relation between the staining properties of bacteria and their relation towards hops antiseptic; Part 3. Journal of the Institute of Brewing, 1937b. 43: p. 191-195.
10. Shimwell, J.L., On the relation between the staining properties of bacteria and their relation towards hops antiseptic; Part 1 + 2. Journal of the Institute of Brewing, 1937a. 43: p. 111-118.
11. Simpson, W.J., Ionophoric action of trans-isohumulone on Lactobacillus brevis. Journal of General Microbiology, 1993b. 139: p. 1041 - 1045.
12. Simpson, W.J., Studies on the sensitivity of lactic acid bacteria to hop bitter acids. Journal of the Institute of Brewing, 1993a. 99(5): p. 405 - 411.
13. Geißler, A., Lifestyle of beer spoiling lactic acid bacteria, in Wissenschaftszentrum Weihenstephan für Ernährung, Landnutzung und Umwelt, Lehrstuhl für Technische Mikrobiologie. 2016, TU München: Freising.
14. Behr, J. and R.F. Vogel, Mechanisms of hop inhibition include the transmembrane redox reaction. Applied and Environmental Microbiology, 2010. 76(1): p. 142 - 149.
15. Kehres, D.G. and M.E. Maguire, Emerging themes in manganese transport, biochemistry and pathogenesis in bacteria. Fems Microbiology Reviews, 2003. 27(2-3): p. 263-290.
16. Back, W., Colour Atlas and Handbook of Beverage Biology Vol. 2. 2005, Nürnberg: Hans Carl Verlag.
17. Behr, J., Mechanisms of hop inhibition, tolerance and adaptation in Lactobacillus brevis in Wissenschaftszentrum Weihenstephan. 2008, Technische Universität München: Freising.
18. Behr, J., M.G. Ganzle, and R.F. Vogel, Characterization of a highly hop-resistant Lactobacillus brevis strain lacking hop transport. Applied and Environmental Microbiology, 2006. 72(10): p. 6483-6492.
19. Bergsveinson, J., et al., Role of plasmids in Lactobacillus brevis BSO 464 hop tolerance and beer spoilage. Applied and Environmental Microbiology, 2015a. 81(4): p. 1234 - 1241.
20. Feyereisen, M., et al., Comparative genome analysis of the Lactobacillus brevis species. Bmc Genomics, 2019c. 20.
21. Iijima, K., et al., HorC confers beer-spoilage ability on hop-sensitive Lactobacillus brevis ABBC45(cc). Journal of Applied Microbiology, 2006. 100(6): p. 1282 - 1288.
22. Preissler, P., Categorization of Lactobacillus brevis along their beer-spoiling potential, in Wissenschaftszentrum Weihenstephan. 2011, Technische Universität München: Freising.
23. Sakamoto, K., et al., Hop resistance in the beer spoilage bacterium Lactobacillus brevis is mediated by the ATP-binding cassette multidrug transporter HorA. Journal of Bacteriology, 2001. 183(18): p. 5371 - 5375.
24. Zhao, Y., S. Knochel, and H. Siegumfeldt, Heterogeneity between and within strains of Lactobacillus brevis exposed to beer compounds. Frontiers in Microbiology, 2017. 8.
25. Behr, J., et al., The identification of novel diagnostic marker genes for the detection of beer spoiling Pediococcus damnosus strains using the BlAst Diagnostic Gene findEr. Plos One, 2016. 11(3).
26. Garcia-Garcia, J.H., et al., Pediococcus damnosus strains isolated from a brewery environment carry the horA gene. Journal of the Institute of Brewing, 2017a. 123(1): p. 77-80.
27. Suzuki, K., et al., Characterization of horA and its flanking regions of Pediococcus damnosus ABBC478 and development of more specific and sensitive horA PCR method. Letters in Applied Microbiology, 2006a. 42(4): p. 392-399.
28. Schneiderbanger, J., F. Jacob, and M. Hutzler, Mini-Review: The current role of lactic acid bacteria in beer spoilage. Brewing Science, 2020. 73: p. 1-5.
29. Schneiderbanger, J., et al., Statistical evaluation of beer spoilage bacteria by real-time PCR analyses from 2010 to 2016. Journal of the Institute of Brewing, 2018. 124(2): p. 173-181.
30. Suzuki, K., 125th Anniversary Review: Microbiological Instability of Beer Caused by Spoilage Bacteria. Journal of the Institute of Brewing, 2011. 117(2): p. 131 - 155.
31. Hayashi, N., et al., Molecular cloning of a putative divalent-cation transporter gene as a new genetic marker for the identification of Lactobacillus brevis strains capable of growing in beer. Applied Microbiology and Biotechnology, 2001. 55(5): p. 596 - 603.
32. Haakensen, M., et al., Ability of novel ATP-binding cassette multidrug resistance genes to predict growth of Pediococcus isolates in beer. Journal of the American Society of Brewing Chemists, 2009d. 67(3): p. 170 - 176.
33. Feyereisen, M., et al., A Plasmid-Encoded Putative Glycosyltransferase Is Involved in Hop Tolerance and Beer Spoilage in Lactobacillus brevis. Applied and Environmental Microbiology, 2020a. 86(3).
34. Bergsveinson, J., et al., Genetic Variability in the Hop-Tolerance horC Gene of Beer-Spoiling Lactic Acid Bacteria. Journal of the American Society of Brewing Chemists, 2016. 74(3): p. 173-182.
35. Vogel, R.F., Preissler, P., Behr, J., Towards an understanding of hop tolerance in beer spoiling Lactobacillus brevis. Brewing Science, 2010. 63: p. 23 - 30.
36. Haakensen, M.C., et al., HorA-specific real-time PCR for detection of beer-spoilage lactic acid bacteria. Journal of the American Society of Brewing Chemists, 2007. 65(3): p. 157 - 165.
37. Suzuki, K., Gram-positive spoilage bacteria in brewing, in Brewing Microbiology - Managing Microbes, Ensuring Quality and Valorising Waste, A.E. Hill, Editor. 2015, Woodhead Publishing: Heidelberg.
38. Sami, M., et al., A plasmid harboring a multidrug resistance-like gene, horA, confers resistance to iso-a-acid in Lactobacillus brevis. Abstracts of the General Meeting of the American Society for Microbiology, 1998. 98: p. 419.
39. Sami, M., et al., Hop-resistant Lactobacillus brevis contains a novel plasmid harboring a multidrug resistance-like gene. Journal of Fermentation and Bioengineering, 1997. 84(1): p. 1-6.
40. Haakensen, M., A. Schubert, and B. Ziola, Multiplex PCR for putative Lactobacillus and Pediococcus beer-spoilage genes and ability of gene presence to predict growth in beer. Journal of the American Society of Brewing Chemists, 2008a. 66(2): p. 63-70.
41. Suzuki, K., et al., A review of hop resistance in beer spoilage lactic acid bacteria. Journal of the Institute of Brewing, 2006c. 112(2): p. 173 - 191.
42. Sami, M., et al., A new and rapid method for determination of beer-spoilage ability of lactobacilli. Journal of the American Society of Brewing Chemists, 1997. 55(4): p. 137-140.
43. Suzuki, K., et al., Isolation of a hop-sensitive variant of Lactobacillus lindneri and identification of genetic markers for beer spoilage ability of lactic acid bacteria. Applied and Environmental Microbiology, 2005a. 71(9): p. 5089 - 5097.
44. Bergsveinson, J., V. Pittet, and B. Ziola, RT-qPCR analysis of putative beer-spoilage gene expression during growth of Lactobacillus brevis BSO 464 and Pediococcus claussenii ATCC BAA-344(T) in beer. Applied Microbiology and Biotechnology, 2012. 96(2): p. 461-470.
45. Bourdineaud, J.P., et al., A bacterial gene homologous to ABC transporters protect Oenococcus oeni from ethanol and other stress factors in wine. International Journal of Food Microbiology, 2004. 92(1): p. 1-14.
46. Waters, L.S., Bacterial manganese sensing and homeostasis. Current Opinion in Chemical Biology, 2020. 55: p. 96-102.
47. Zheng, F.Y., et al., Roles of Divalent-Cation Transporter Genes mntB and mntC of Beer Spoilage Bacteria in Resisting Hop Bitter Compound Iso-alpha-Acid. Journal of the American Society of Brewing Chemists, 2020.
48. Feyereisen, M., et al., Beer spoilage and low pH tolerance is linked to manganese homeostasis in selected Lactobacillus brevis strains. Journal of Applied Microbiology, 2020b. 129(5): p. 1309-1320.
49. Dols-Lafargue, M., et al., Characterization of gtf, a glucosyltransferase gene in the genomes of Pediococcus parvulus and Oenococcus oeni, two bacterial species commonly found in wine. Applied and Environmental Microbiology, 2008. 74(13): p. 4079 - 4090.
50. Pittet, V., K. Morrow, and B. Ziola, Ethanol tolerance of lactic acid bacteria, including relevance of the exopolysaccharide gene gtf. Journal of the American Society of Brewing Chemists, 2011. 69(1): p. 57 - 61.
51. Suzuki, K., et al., Biochemical characterization of horA-independent hop resistance mechanism in Lactobacillus brevis. International Journal of Food Microbiology, 2002. 76(3): p. 223-230.
52. Behr, J., et al., Identification of ecotype-specific marker genes for categorization of beer-spoiling Lactobacillus brevis. Food Microbiology, 2015. 51: p. 130 - 138.
| Name | Datei |
|---|---|
| Auf die Gene kommt es an |
 Download Download
|